

De nouvelles souches de grippe porcine circulent « inaperçues » chez les porcs au Cambodge (Image : Getty Images)
Il a été constaté que plusieurs souches jusque-là inconnues de la grippe porcine circulaient « inaperçues » chez les porcs au Cambodge depuis des années, ce qui pose un risque potentiel de pandémie.
C’est l’avertissement d’une équipe internationale de chercheurs, qui a noté que certaines des nouvelles souches semblent contenir des virus transmis aux porcs par l’homme.
L’équipe a expliqué que les porcs sont un intermédiaire clé dans l’émergence et la propagation potentielle des virus de la grippe entre les animaux et les humains.
Les porcs fournissent un environnement approprié pour le brassage de segments génétiques entre les hôtes aviaires, porcins et humains, permettant ainsi l’évolution de nouveaux virus.
Ce risque a été accru, notent les experts, par l’augmentation de la production porcine au cours des cinq dernières décennies, qui a accru les déplacements et le commerce international des animaux.
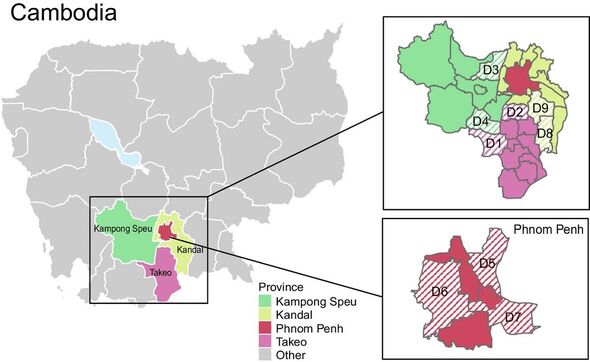
L’équipe a échantillonné des porcs dans 18 abattoirs répartis dans quatre provinces cambodgiennes. (Image : Zeller et al. / PNAS)
L’enquête a été entreprise par le professeur Yvonne Su, biologiste évolutionniste, de la faculté de médecine de l’Université nationale Duke de Singapour, et ses collègues.
Su a déclaré : « L’évolution à long terme de différentes lignées a conduit à l’établissement de virus génétiquement distincts qui circulent continuellement dans les populations porcines sans être détectés depuis des décennies.
« Notre étude a révélé le paysage génomique caché et complexe de l’évolution du virus de la grippe porcine en Asie du Sud-Est. »
Cette région, a-t-elle ajouté, est considérée « comme un point chaud en termes de diversité virale et de risque d’émergence de nouveaux virus ».
L’équipe a découvert que le sous-type H1N1 (représenté) était prédominant et provenait probablement de l’homme. (Image : Getty Images)
Dans leur étude, l’équipe a mené une surveillance de la grippe porcine dans 18 abattoirs de porcs au Cambodge entre mars 2020 et juillet 2022.
Au total, ils ont collecté plus de 4 000 écouvillons nasaux sur des porcs répartis dans quatre provinces différentes – parmi lesquels 72 animaux, soit environ deux pour cent de ceux testés, se sont révélés positifs pour le virus de la grippe A.
Parmi ces virus, Su et ses collègues ont identifié neuf groupes différents de grippe porcine A, dont au moins sept n’avaient pas été détectés auparavant depuis deux à 15 ans.
L’équipe a découvert plusieurs lignées H3 qui avaient été transmises aux porcs par les humains – et semblent circuler sans être détectées depuis environ une décennie.
Ils rapportent également que le sous-type H1N1 était prédominant et probablement dérivé de l’homme, remontant à la pandémie mondiale de 2009.
Chez les porcs des provinces de Kandal, Phnom Penh et Takeo, l’étude a identifié deux virus saisonniers qui proviendraient de Thaïlande.
Les chercheurs rapportent également avoir isolé une nouvelle variante porcine européenne du H1N2 – dérivée à l’origine d’oiseaux – dont les gènes remontent à l’Amérique du Nord.
Bien qu’elle ait été la première à détecter ce variant, l’analyse génomique suggère qu’il circule sans être détecté chez les porcs au Cambodge depuis 2014.
Des preuves génétiques indiquent que les virus de la grippe porcine européenne semblent avoir été introduits sporadiquement en Chine et en Asie du Sud-Est au début des années 2000.
En outre, le centre-sud de la Chine semble avoir été la principale source de transmission du virus de la grippe porcine de type européen dans la région depuis 2010, ces souches se propageant plus largement à travers la Chine, puis dans les pays d’Asie du Sud-Est comme le Cambodge.
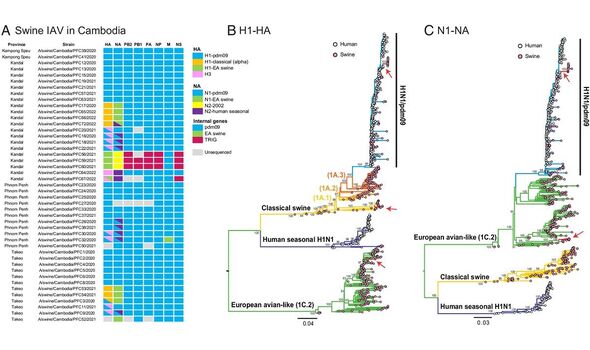
Su et ses collègues ont identifié neuf groupes différents de grippe porcine A (Image : Zeller et al. / PNAS)
Le professeur Gavin Smith, biologiste évolutionniste, est co-auteur de l’étude et directeur du programme Duke-NUS Emerging Infectious Diseases.
Il a déclaré : « Bien que les virus de la grippe porcine provoquent généralement de légers symptômes chez les porcs, ils constituent une menace pandémique pour les humains.
« Il se peut que la population humaine manque d’immunité ou soit insuffisamment protégée contre les nouvelles souches de virus de la grippe porcine.
« Par conséquent, une surveillance systématique est cruciale pour la détection précoce et l’avertissement de nouveaux sous-types ou souches. »
Une fois leur étude initiale terminée, les chercheurs travaillent désormais au développement d’une plateforme capable d’identifier tous les principaux sous-types génétiques de la grippe porcine, ce qui, espèrent-ils, aidera à mieux évaluer la propagation et la menace pandémique des nouveaux virus.
Cet examen, ont-ils expliqué, inclura non seulement les sous-types porcins et humains, mais également plusieurs séquences aviaires, leur permettant d’identifier si les populations porcines et humaines ont été infectées par les sous-types de grippe.
Le professeur Patrick Tan — vice-doyen principal à la recherche de Duke-NUS — a déclaré : « Une surveillance de routine et soutenue est indispensable pour identifier de nouveaux virus afin que leur risque de transmission puisse être évalué.
« Il est donc essentiel que des méthodes de surveillance plus efficaces et continues soient intégrées à des outils analytiques automatisés pour fournir rapidement des informations sur les changements dans les agents pathogènes humains et animaux.
« Un système tel que celui que développe l’équipe de Duke-NUS améliorerait la santé animale grâce à la sélection de vaccins efficaces et contribuerait à la santé humaine en surveillant les virus susceptibles de se transmettre. »
Les résultats complets de l’étude ont été publiés dans la revue PNAS.



